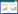2. 郑州大学第一附属医院急诊医学部,河南省中毒类疾病医学重点实验室,郑州 450000;
3. 武汉大学生命科学学院,病毒学国家重点实验室,武汉 430072;
4. 郑州大学生命科学院,郑州 450001
脓毒症是由感染引起的宿主反应失调导致的危及生命的器官功能障碍[1-2]。全球每年影响数百万人,既往研究显示脓毒症病死率约26.7%[3]。在引发脓毒症的各类感染中,血流感染占比约40%[4]。在脓毒症患者的救治中,尽早合理使用抗感染药物对其预后至关重要[2, 5]。因此亟需一种快速而精准的病原学诊断方法为抗感染药物方案的合理制定提供病原学依据。“血培养”目前仍为血流感染诊断的金标准,该方法检测周期长、阳性检出率低及病原体检测普窄等缺点不可忽视。随着宏基因组二代测序技术(metagenomic next-generation sequencing,mNGS)在感染性疾病中普及应用[6],其快速、无偏倚、广覆盖的优点补充了传统病原学检测方法的不足,且能进行病原体定量分析以指导抗感染药物疗程。为探讨mNGS在血流感染脓毒症患者诊疗中的应用价值,本研究回顾分析本院同时行血液mNGS和血培养检测的脓毒症患者资料,对比分析两种检测方法对血流感染脓毒症的诊断效能,并评估mNGS对抗感染方案制定的影响。
1 资料与方法 1.1 一般资料选取本院2020年1月至2021年9月收治的脓毒症且考虑血流感染的患者。纳入标准:(1)符合2016国际脓毒症指南诊断标准[2];(2)同时行血培养和血液mNGS检测的患者(送检血培养和mNGS时间在24 h内视为同时送检);(3)在住院期间根据临床表现和实验室检查,高度怀疑存在血流感染,符合以下条件:体温 > 38℃或 < 36℃,可伴有寒战或全身中毒症状等,并合并下列情况之一:①有病原体入侵部位或迁徙病灶。②有全身中毒症状而无明显感染灶。③有皮疹或出血点、肝脾肿大、血中性粒细胞增多伴核左移且无其他原因可解释。④收缩压低于90 mmHg (1 mmHg=0.133 kPa)或下降超过40 mmHg[7];⑤年龄 > 18岁。排除标准:①临床病历资料不完整;②孕产妇。本研究已在本院伦理委员会备案并通过审批(审批号2021-KY-0953-001),该研究为回顾性研究,患者个人信息保密,故无需签署知情同意书。
1.2 研究方法收集患者的一般资料[年龄、性别、送检mNGS当天序贯器官衰竭(SOFA)评分]、检验结果(血常规、肝肾功能、降钙素原、凝血功能)、临床参数(体温、心率、平均动脉压、血管活性药物使用与否、机械通气与否、血液净化与否、激素应用与否)、病原学检测结果(血培养结果、mNGS结果)、预后(生存或死亡)。若同一患者同次住院期间,有二次或多次复查的血液mNGS和血培养结果,符合条件均纳入分析。
1.3 统计学方法采用SPSS 25.0软件进行统计学分析,采用Kolmogorov-Smirnov法对计量资料进行正态性检验,正态分布以均数±标准差(x±s)表示,非正态分布以中位数及四分位数(Inter Quartile Range, IQR)表示;计数资料以例数及百分比(n, %)表示;率的比较采用χ2检验;一致性检验以Kappa值表示。以P<0.05为差异有统计学意义。
2 结果 2.1 基本资料本研究共纳入107例患者,其中85例患者送检mNGS及血培养1次,19例患者送检mNGS及血培养2次,3例患者送检mNGS及血培养3次,共分析132份血液mNGS及血培养结果。样本纳入及对比分析流程图见图 1,患者的临床特征见表 1。

|
| 图 1 样本纳入及对比分析流程图 |
|
|
| 临床特征 | 患者(n=107) |
| 年龄(x±s,岁) | 53.41±18.57 |
| SOFA评分[M(IQR)] | 6(3,10) |
| 男性(n,%) | 63(58.9) |
| 女性(n,%) | 44(41.1) |
| 实验室检查结果 | |
| 白细胞计数[M(IQR),×109/L] | 9.30(4.88,15.20) |
| 血小板计数[M(IQR),×106/L] | 107(40,210) |
| 降钙素原[M(IQR),ng/dL] | 1.29(0.41,5.50) |
| 肌酐[M(IQR),mmol/L] | 79(46,138) |
| 总胆红素[M(IQR),mmol/L] | 12.50(8.70,24.00) |
| 凝血酶原时间[M(IQR),s] | 13.50(11.80,15.30) |
| 体温[M(IQR),℃] | 38.00(37.30,38.50) |
| MAP(x±s,mmHg) | 87.61±13.59 |
| 临床治疗情况(n,%) | |
| 使用血管活性药物 | 43(40.19) |
| 使用机械通气 | 40(37.38) |
| 使用血液净化 | 17(15.89) |
| 使用激素 | 27(25.23) |
| 预后(n,%) | |
| ICU病死率 | 23(17.42) |
| 住院病死率 | 23(17.42) |
mNGS共检出51种病原体,其中细菌24种、真菌8种、病毒17种、特殊病原体2种。血培养共检出15种病原体,其中细菌13种、真菌2种。mNGS检出116株细菌(G-菌71.6%,G+菌28.4%),检出频次前5位为肺炎克雷伯菌、屎肠球菌、大肠埃希菌、铜绿假单胞菌、鲍曼不动杆菌;血培养检出26株细菌(G-菌77%,G+菌23%),检出频次前3位为肺炎克雷伯菌、大肠埃希菌、鲍曼不动杆菌。mNGS检出23株真菌,检出频次前3位为烟曲真菌、热带念珠菌、小孢根霉;血培养检出2株热带念珠菌、1株白色念珠菌。见图 2。mNGS检出73株病毒,检出频次前3位为巨细胞病毒、EBV病毒、人类疱疹病毒1型。此外,mNGS还检出1株结核分枝杆菌、1株人型支原体。

|
| 图 2 mNGS和血培养检测细菌、真菌病原体的结果 |
|
|
在总病原体检出阳性率方面,mNGS显著高于血培养,两者比较,差异有统计学意义(85.6% vs. 18.2%,P < 0.01)。在细菌及真菌检出阳性率方面,mNGS均高于血培养,差异均有统计学意义(P < 0.01)。以血培养为“金标准”,mNGS诊断血流感染的灵敏度为95.8%、特异度为16.7%、阳性预测值为20.3%、阴性预测值为94.7%。其中mNGS和血培养均为阳性者占17.4%,仅mNGS为阳性占68.2%,仅血培养为阳性占0.8%。Kappa一致性测试显示这两种方法一致性低(k=0.051,P=0.115)。见表 2、3。
| 方法 | 血培养(+) | 血培养(-) | 合计 | 一致性检验 | |
| Kappa值 | P值 | ||||
| mNGS(+) | 23 | 90 | 113 | 0.051 | 0.115 |
| mNGS(-) | 1 | 18 | 19 | ||
| 合计 | 24 | 108 | 132 | ||
| 注:灵敏度=95.8%,特异度=16.7%,阳性预测值=20.3%,阴性预测值=94.7% | |||||
| 样本 | mNGS检出阳性率(%) | 血培养检出阳性率(%) | χ2值 | P值 |
| 总体病原体 | 85.6 | 18.2 | 120.19 | < 0.01 |
| 细菌 | 63.6 | 15.9 | 62.76 | < 0.01 |
| 真菌 | 13.6 | 2.3 | 11.64 | < 0.01 |
根据132份mNGS结果,42.4%(n=56)的抗感染方案进行调整。共有25份为复测的mNGS,初测和复测间隔平均天数为9 d,根据复测的mNGS结果,40%(n=10)的抗感染方案进行调整。见表 4。
| mNGS对抗感染方案制定的影响 | 总体132份mNGS | 复测的25份mNGS |
| 调整 | ||
| 抗感染方案升级 | 41/132(31.1) | 6/25(24) |
| 抗感染方案降级 | 5/132(3.8) | 2/25(8) |
| 抗感染方案停用 | 2/132(1.5) | 2/25(8) |
| 停用不必要药物+未覆盖药物 | 4/132(3.0) | |
| 加用未覆盖药物 | 4/132(3.0) | |
| 未调整 | ||
| 证实临床诊断并维持原抗感染方案 | 36/132(27.3) | 8/25(32) |
| 没有明确致病菌 | 35/132(26.5) | 6/25(24) |
| 明确致病菌但家属拒绝调整用药 | 4/132(3.0) | 1/25(4) |
| 明确致病菌但患者死亡 | 1/132(0.8) |
随着mNGS技术不断进步与发展,其应用于临床病原学诊断方面日趋成熟。本研究回顾分析132份血流感染脓毒症患者的mNGS与血培养结果,发现mNGS具有较高的灵敏度和阳性检出率,且mNGS检出总病原体的种类及数量高于血培养;此外mNGS为42.4%的抗感染方案优化调整提供病原学依据。在病原体检测结果方面,mNGS检出的病原体以细菌、病毒为主,其次为真菌、特殊病原体,这一结果与既往文献结果基本一致[8-10]。mNGS除发现细菌、真菌以外,还能同时发现病毒、特殊病原体感染。血培养无法检测病毒,本研究中mNGS检出73株病毒,病毒阳性检出率较高(57/132,43.2%)。目前临床常用的病毒检测方法有酶联免疫吸附试验、聚合酶链式反应等,但只能检出限定的病毒,且受临床医生对病情判断的影响[9]。能同时检出细菌、真菌、病毒和特殊病原体等混合感染,是mNGS的一大优势[6]。
对比血培养结果,mNGS灵敏度为95.8%、特异度为16.7%,mNGS与血培养阳性检出率分别为85.6% vs. 18.2%,差异有统计学意义(P < 0.01)。笔者发现mNGS在病原体检测方面有较高的灵敏度和阳性检出率,且检出病原体类别及数量较血培养显著增多。Grumaz等[8]的研究纳入239例脓毒症患者,通过mNGS检出74种病原体,而血培养仅检出13种病原体,本研究也观察到类似的现象,mNGS检出病原体种类远高于血培养。但mNGS的特异性较低,且两种检测方法检出结果的一致性强度低,本研究分析可能有以下原因:①以血培养作为金标准,但血培养的阳性率低,本研究中为18.2%,与既往研究结果相似(10%~50%)[11-12];②血培养无法检测病毒,在本研究中有14份样本(10.6%)mNGS仅检出病毒感染,而血培养为阴性;③先前使用抗菌药物对血培养的影响可能大于对mNGS影响[13],本院为省级医院,患者来源以下级医院转诊为主,大多数患者在外院已接受经验性抗感染治疗,这可能会是血培养阳性率低的原因之一。④关于mNGS报告的临床解读仍存在很大的困难和挑战[14],目前尚缺乏国际公认的判读标准,本研究中mNGS结果判读可能较为宽松,例如当mNGS结果提示某病原体序列数和相对丰度较低时,其是否为致病菌仍需结合临床,但本研究将其结果直接判读为阳性,可能会增加mNGS阳性检出率。综上,笔者认为mNGS的灵敏度和阳性检出率是较高的,但当与传统培养结果不一致时仍需临床医生结合病情综合判断。
对治疗方案制定的影响方面,Zhou等[15]的研究发现mNGS使37.1%患者的治疗方案调整,其中包括25.2%患者的抗感染方案降级。与之相似,本研究中42.4%的抗感染方案是依据mNGS结果进行调整。然而Hogan等[16]研究发现mNGS对大多数患者(92.7%)没有影响或产生负面影响(如mNGS致不必要的治疗、增加额外检查、延长住院时间、对诊疗方案无影响等),其原因可能是其纳入的研究对象为某时间段所有送检血液mNGS的患者,而本研究仅针对血流感染的脓毒症患者,入组患者不同可能导致两者结论差异。Grumaz等[17]提出了脓毒症指数概念,脓毒症患者一旦开启有效的抗感染治疗,其指数会显著下降。利用mNGS定量分析病原体序列数的优点,可以及时优化抗感染方案。Niles等[18]的研究对37例某时间段送检血液mNGS的患儿进行了mNGS的复测,其中7%(4/37)的病例依据mNGS调整抗感染方案。本研究有25份mNGS复测结果,其中40%(n=10)的抗感染方案依据mNGS进行调整,该比例高于既往研究,可能原因为Niles等的研究初测和复测间隔平均天数为47 d,而本研究中初测和复测间隔平均天数为9 d,短期内复测可及时发现患者致病菌的变化,利用其定量分析优势比较治疗前后病原体序列数变化从而及时优化抗感染方案。
利益冲突 所有作者声明无利益冲突
作者贡献声明 韦毅萌、袁丁:实施研究、分析/解释数据、论文撰写;余言午、徐志高、车璐:统计分析、支持性贡献;刘映乐、张守涛、田庆南:技术支持、指导;侯林林、张岩、孟翠翠、杨放、闫弘义、杜宇琪:数据收集及整理;高艳霞:研究设计、论文修改、技术支持、获取研究经费
| [1] | 齐文旗, 张斌, 郑忠骏, 等. 拯救脓毒症运动: 2021年国际脓毒症和脓毒性休克管理指南[J]. 中华急诊医学杂志, 2021, 30(11): 1300-1304. DOI:10.3760/cma.j.issn.1671-0282.2021.11.003 |
| [2] | Rhodes A, Evans LE, Alhazzani W, et al. Surviving Sepsis campaign: international guidelines for management of sepsis and septic shock: 2016[J]. Intensive Care Med, 2017, 43(3): 304-377. DOI:10.1007/s00134-017-4683-6 |
| [3] | Fleischmann-Struzek C, Mellhammar L, Rose N, et al. Incidence and mortality of hospital- and ICU-treated sepsis: results from an updated and expanded systematic review and meta-analysis[J]. Intensive Care Med, 2020, 46(8): 1552-1562. DOI:10.1007/s00134-020-06151-x |
| [4] | Timsit JF, Ruppé E, Barbier F, et al. Bloodstream infections in critically ill patients: an expert statement[J]. Intensive Care Med, 2020, 46(2): 266-284. DOI:10.1007/s00134-020-05950-6.4 |
| [5] | 中国医师协会急诊医师分会, 中国研究型医院学会休克与脓毒症专业委员会. 中国脓毒症/脓毒性休克急诊治疗指南(2018)[J]. 临床急诊杂志, 2018, 19(9): 567-588. DOI:10.13201/j.issn.1009-5918.2018.09.001 |
| [6] | Chiu CY, Miller SA. Clinical metagenomics[J]. Nat Rev Genet, 2019, 20(6): 341-355. DOI:10.1038/s41576-019-0113-7 |
| [7] | 中华人民共和国卫生部. 医院感染诊断标准(试行)[J]. 中华医学杂志, 2001, 81(5): 314-320. DOI:10.3760/j:issn:0376-2491.2001.05.027 |
| [8] | Grumaz S, Grumaz C, Vainshtein Y, et al. Enhanced performance of next-generation sequencing diagnostics compared with standard of care microbiological diagnostics in patients suffering from septic shock[J]. Crit Care Med, 2019, 47(5): e394-e402. DOI:10.1097/CCM.0000000000003658 |
| [9] | Long Y, Zhang YX, Gong YP, et al. Diagnosis of sepsis with cell-free DNA by next-generation sequencing technology in ICU patients[J]. Arch Med Res, 2016, 47(5): 365-371. DOI:10.1016/j.arcmed.2016.08.004 |
| [10] | Jing CD, Chen HB, Liang Y, et al. Clinical evaluation of an improved metagenomic next-generation sequencing test for the diagnosis of bloodstream infections[J]. Clin Chem, 2021, 67(8): 1133-1143. DOI:10.1093/clinchem/hvab061 |
| [11] | Burillo A, Bouza E. Use of rapid diagnostic techniques in ICU patients with infections[J]. BMC Infect Dis, 2014, 14: 593. DOI:10.1186/s12879-014-0593-1 |
| [12] | Previsdomini M, Gini M, Cerutti B, et al. Predictors of positive blood cultures in critically ill patients: a retrospective evaluation[J]. Croat Med J, 2012, 53(1): 30-39. DOI:10.3325/cmj.2012.53.30 |
| [13] | Miao Q, Ma YY, Wang QQ, et al. Microbiological diagnostic performance of metagenomic next-generation sequencing when applied to clinical practice[J]. Clin Infect Dis, 2018, 67(suppl_2): S231-S240. DOI:10.1093/cid/ciy693 |
| [14] | Chiang AD, Dekker JP. From the pipeline to the bedside: advances and challenges in clinical metagenomics[J]. J Infect Dis, 2020, 221(Suppl 3): S331-S340. DOI:10.1093/infdis/jiz151 |
| [15] | Zhou H, Larkin PMK, Zhao DD, et al. Clinical impact of metagenomic next-generation sequencing of bronchoalveolar lavage in the diagnosis and management of pneumonia: a multicenter prospective observational study[J]. J Mol Diagn, 2021, 23(10): 1259-1268. DOI:10.1016/j.jmoldx.2021.06.007 |
| [16] | Hogan CA, Yang SX, Garner OB, et al. Clinical impact of metagenomic next-generation sequencing of plasma cell-free DNA for the diagnosis of infectious diseases: a multicenter retrospective cohort study[J]. Clin Infect Dis, 2021, 72(2): 239-245. DOI:10.1093/cid/ciaa035 |
| [17] | Grumaz S, Stevens P, Grumaz C, et al. Next-generation sequencing diagnostics of bacteremia in septic patients[J]. Genome Med, 2016, 8(1): 73. DOI:10.1186/s13073-016-0326-8 |
| [18] | Niles DT, Revell PA, Ruderfer D, et al. Clinical impact of plasma metagenomic next-generation sequencing in a large pediatric cohort[J]. Pediatr Infect Dis J, 2022, 41(2): 166-171. DOI:10.1097/INF.0000000000003395 |
 2023, Vol. 32
2023, Vol. 32